运行结束后输出密度rho
-
@wangfei9088 好的好的,谢谢前辈!!
-
@wangfei9088 前辈这个我之前说的有点问题,我之前以为敏感性分析就是分析每个基元反应的速率,结果好像不是!我目前就是想得到计算收敛后每个基元反应的速率,然后和流场结果对比一下,确定在哪块流场区域哪个基元反应相对更重要


-
@风云5091 我的知识也有限,没见过后处理有这个计算基元反应速率的功能。提供个思路。
StandardChemistryModel.H 里有计算某个反应中某个组分反应速率的函数://- Return reaction rate of the speciei in reactionI virtual tmp<volScalarField::Internal> calculateRR ( const label reactionI, const label speciei ) const;specieReactionRates.C 用到了上面那个函数,得到的是总的反应速率:
volScalarField::Internal RR ( chemistryModel_.calculateRR(ri, si) );我想应该可以仿照它写一个满足要求的functionObjects吧。供参考。
-
@wangfei9088 好的,感谢前辈提供的思路,基元反应速率在我研究的这个领域还挺重要的,至少我看的大部分文献里都提到了这个,暑假可能要多学习学习openfoam代码了


-
@wangfei9088 如果可以的话,前辈您可以提供一些您当时学习燃烧这块openfoam源代码的思路/资料吗,感觉这块李老师研究的也比较少,网上能搜索到的资源也比较少。
-
@wangfei9088 好的好的,谢谢前辈!!


-
大佬,我跑个题。我看你那面做化学反应比较多。有没有兴趣用PINN搞。化学反应用PINN,其他部分还是用传统CFD。
这个路子你们组里面搞了没。没搞的话感兴趣搞么。 -
@wangfei9088 请教大佬,参照specieReactionRates的写法,先尝试输出每个反应步的体积平均Qdot(命名sumVQdotRi/V),计算Qdot时需要物种的比焓h[speciei],这个如何访问呢?
const scalar h[speciei] = specieThermos_h[speciei].hf(); //如何访问?
bool Foam::functionObjects::reactionSpecieQdots::write() { logFiles::write(); const label nSpecie = chemistryModel_.nSpecie(); const label nReaction = chemistryModel_.nReaction(); // Region volume const scalar V = this->V(); for (label reactioni=0; reactioni<nReaction; reactioni++) { if (Pstream::master()) { writeTime(file()); file() << token::TAB << reactioni; } const PtrList<volScalarField::Internal> RR ( chemistryModel_.reactionRR(reactioni) ); scalar sumVQdotsR = 0; for (label speciei=0; speciei<nSpecie; speciei++) { scalar sumVQdotRi = 0; const scalar h[speciei] = specieThermos_h[speciei].hf(); //如何访问? if (all()) { sumVQdotRi = fvc::domainIntegrate(RR[speciei]*(-h[speciei])).value(); } else { sumVQdotRi = gSum ( scalarField ( fvMeshFunctionObject::mesh_.V()*RR[speciei]*(-h[speciei]), cells() ) ); } if (Pstream::master()) { file() << token::TAB << sumVQdotRi/V; } sumVQdotsR += sumVQdotRi; } if (Pstream::master()) { file() << token::TAB << sumVQdotsR/V; } if (Pstream::master()) { file() << nl; } } if (Pstream::master()) { file() << nl << endl; } return true; } -
@wangfei9088 这是我看到基金会版本里面用来算Qdot的,我直接贴上去了。我的问题是specieThermo_这个可以直接访问吗?const label nSpecie = chemistryModel_.nSpecie();可以这么写是前面定义了
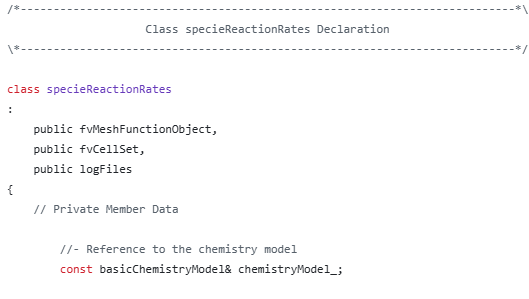
我看到specieThermo_是在别的地方定义的。我弄不明白这个。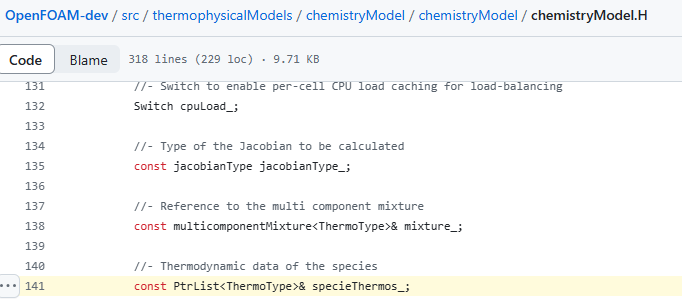
-
@尚善若水 看了一下,开发版和of2012差别很大,开发版里不能用specieThermo_了。依据要不要单位,组分的生成焓在开发版里可以这么写:
dimensionedScalar hi = chemistryModel_.thermo().hfi(speciei); //- Enthalpy of formation [J/kg] 或者 scalar hi = chemistryModel_.thermo().hfiValue(speciei); //- Enthalpy of formation [J/kg] -
@wangfei9088 好的,我仔细看下。谢谢。
-
@wangfei9088 感谢大佬,程序正常输出,正确性还没测试,也许可以和cantera的结果比一下,代码就是上面发的加上根据大佬说的改的。另外,我在想可不可以更进一步,写出每一个反应步每个组分的Qdot,命名为Qdot.Ri.Speciei,类似输出RR.Speciei?
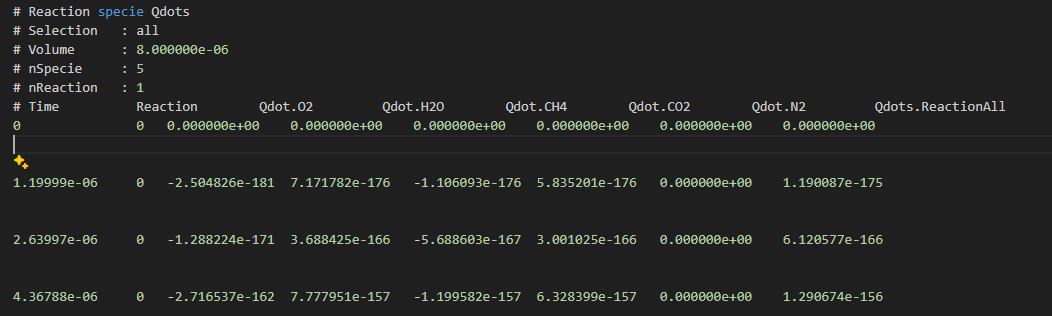
-
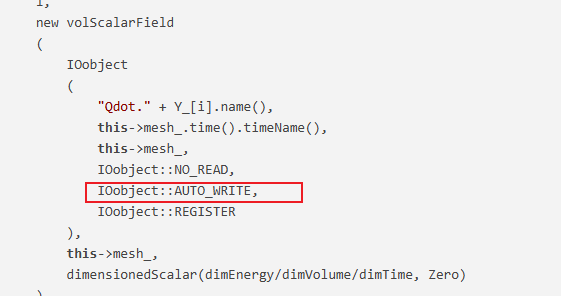
对于你的问题,我最近也在修改,加入以下代码,然后在类定义中添加成员变量就行了。我这边是可以成功输出每个物质对应的Qdot
template<class ReactionThermo, class ThermoType> Foam::tmp<Foam::volScalarField> Foam::StandardChemistryModel<ReactionThermo, ThermoType>::Qdot() const { tmp<volScalarField> tQdot ( new volScalarField ( IOobject ( "Qdot", this->mesh_.time().timeName(), this->mesh_, IOobject::NO_READ, IOobject::NO_WRITE, IOobject::NO_REGISTER ), this->mesh_, dimensionedScalar(dimEnergy/dimVolume/dimTime, Zero) ) ); QdotSpecies_; if (!QdotSpecies_.size()) { // 首次调用时初始化 QdotSpecies_.setSize(nSpecie_); forAll(Y_, i) { QdotSpecies_.set ( i, new volScalarField ( IOobject ( "Qdot." + Y_[i].name(), this->mesh_.time().timeName(), this->mesh_, IOobject::NO_READ, IOobject::AUTO_WRITE, IOobject::REGISTER ), this->mesh_, dimensionedScalar(dimEnergy/dimVolume/dimTime, Zero) ) ); } } if (this->chemistry_) { scalarField& Qdot = tQdot.ref(); Qdot = 0.0; forAll(Y_, i) { volScalarField& QdotI = QdotSpecies_[i]; const scalar hi = specieThermo_[i].Hc(); forAll(Qdot, celli) { QdotI[celli] = -hi*RR_[i][celli]; Qdot[celli] += QdotI[celli]; } // 修正边界条件 QdotSpecies_[i].correctBoundaryConditions(); } tQdot.ref().correctBoundaryConditions(); } return tQdot; }